Python(1) PyCallGraphを利用したコードプロファイリングの可視化
最近Pythonを使って、特定のmicroRNAが結合するSeed配列をmRNAの配列中から探索して、さらに種間保存性や配列の特徴を解析するスクリプトを思いつくままに書いたのですが、
実行してみて、そのスクリプトがやたら遅いことに気づきました(-_-;)
「1つのmiRNA vs すべてのmRNA」のペアで3時間以上かかる…。すべてのmiRNAとすべてのmRNAの組み合わせを実行したら、いったい計算に何ヶ月かかるんだろうってレベルです。
スクリプトはこちら。
そこで、スクリプトのどの部分が計算上ボトルネックになっているか調べてみることにしました。POSTDに掲載されていたPythonにおけるコードプロファイリングのお話があったので早速試してみました。
上記の記事でも説明されているように、 PythonにはcProfile (pythonのcProfileを使ってみる。 - malibu-bulldogの日記)と呼ばれるプロファイリングツールが用意されています。しかし、実行してみるとログがだぁーと出力されてくるだけで、何を言っているのか直感的に理解できません…。
そこで、PyCallGraphの出番です。PyCallGraphは上記のcProfileの出力情報を視覚化すてくれます。もちろん、ビルトインモジュールではないので、自身でインストールを行う必要があります。
(1) PyCallGraphのインストール
PythonのモジュールであるPyCallGraphとGraphvizをそれぞれインストールします(今回はPython3のモジュールとしてインストールしました)。
また、グラフを描写するために必要となるGraphviz自体がインストールされていない場合は、合わせてインストールする必要があります。当たり前ですが、GraphvizがないとPyCallGraphが使えません(実行するとエラーが返ってくる)。
(2) PyCallGraphの実行
下記の通り、pycallgraphからスクリプトを呼び出すだけでOKです。簡単ですね。
(3) 実行結果
結果としてpycallgraph.pngというファイルがカレントディレクトリに生成されます(この時、同じ名前のファイルが既に存在しているとエラーを起こす原因になるかもしれません)。 出力された画像ファイル(.png)の一部を切り取ってきたのが下図になります。
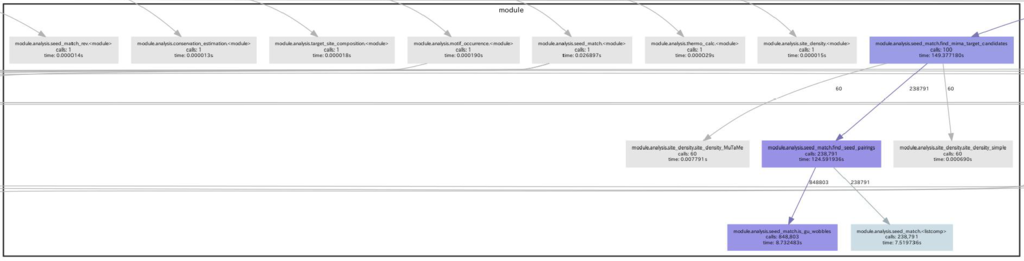
細かくて字がつぶれていますが、「module.analysis.seed_match.find_mirna_target_candidate」モジュールとそれに従属するモジュール群が青色になっています(それ以外は灰色)。
これは、これらのモジュール上で計算に時間がかかっていることを示しています(計算上のボトルネック)。以上の結果から、seed配列の探索に時間がかかっていることが判明しました(計算時間のほとんどがseed配列の探索に割かれている…)。
このようにPyCallGraphを利用したコードプロファイリングを行うことで、スクリプトのどの個所に計算上のボトルネックが存在しているのか視覚的に把握することができます。