BioLinux/Ubuntu(4) 公開鍵認証を利用したSSHによるリモート接続(Windows10=>BioLinux8)
普段、Windows10が入ったノートパソコンを利用していて、解析を行うためにLinuxの入ったデスクトップパソコンを使うことがあります。
恥ずかしい話ですが、今までデータのやり取りをパソコン間で行うために、外付けのハードディスクを使って行っていました。
そういった場合に、片方のパソコンにリモート接続してパソコン間でデータのやり取りが自在にできたら便利だなぁと思っていました。
そんなこんなで今回は、SSHを利用したリモート接続の方法を試してみたいと思います。内容としてはこちらのブログを主に参考にしました。
ubuntuを使ってみた: windows7からubuntu11.10にsshでアクセスしてみる
SSHとは、パスワードや公開鍵認証を利用することで安全にリモートからパソコンに接続するための方法のことです(詳しくは下記を参照)。今回は、よりセキュアな公開鍵を利用した認証を試したいと思います。
SSHは、インフラエンジニア以外にとって馴染みのない技術というわけではなく、いろんなところで利用されています。例えば、次世代シーケンサーのデータをSRAにアップロードする際に利用される接続に、公開鍵認証によるSSHが使われています。
前置きはこのくらいにして、早速設定を行ってみましょう。
使ったもの
- BioLinux8の入ったパソコン(Desktop)
- Windows10の入ったパソコン(Laptop)
公開鍵と秘密鍵の準備
SSHによるリモート接続を行うために、公開鍵と秘密鍵を用意する必要があります。公開鍵はデータの暗号化に使う鍵であり、秘密鍵は暗号化されたデータを復号化する(データを読める形に変換する)のに必要な鍵です(詳しくは下記を参照)。
今回の例では、公開鍵は遠隔操作を受ける側であるBioLinux8の入ったパソコンに、秘密鍵はリモート接続を行う側であるWindows10の入ったパソコンがそれぞれ保持する形にします。
Windows10側の操作
(1) Tera Termのインストール
下記のリンクから、TeraTermをまずインストールします。EXEファイルなのでインストールは簡単。
(2) SSH鍵の生成
TeraTermのインストールが終わったら、TeraTermを起動しましょう。メニューから、設定→SSH鍵生成をクリックします。
「TTSSH: 鍵生成」というウインドウが出てくるので、「生成」ボタンをクリックし、公開鍵と秘密鍵を生成します。このとき、「鍵のパスフレーズ」も設定しておきましょう。そうしたら、公開鍵と秘密鍵をそれぞれ任意の場所に保存してください。
Linuxのパソコンへ公開鍵を入れる必要があるので、USBメモリなどにデータを保存しておきましょう。それでは、BioLinux8側の設定に移りましょう。
BioLinux8側の設定
(1) OpenSSHのインストール
まず、SSH接続を行うためにOpenSSHをインストールします。LinuxではSSHを使う上でOpenSSHはデファクトスタンダードとなっており、BioLinux8にはあらかじめインストールされているはずです。もし万が一、インストールされていない場合は下記のコマンドを実行して、パソコンにOpenSSHをインストールしておきましょう。
下記のコマンドを実行して、SSH鍵を生成します。
(3) Windows10で生成した公開鍵をLinux側の公開鍵ファイルに保存
BioLinux8側にある公開鍵ファイル(~/.ssh/authorized_keys)に、先ほどWindows10上で作成した公開鍵を組み込みます。
下記のコマンドを実行。
(4) SSHの設定をいじくる
下記のコマンドを実行して、
ChallengeResponseAuthentication no
PasswordAuthentication no
UsePAM no
という設定に変更する。「#」が先頭についている場合は削除する。
こうすることで、パスワードによる認証ではなく、SSH鍵による認証によってリモートできるように設定します。
(5) SSHの設定を適応する
下記のコマンドを実行してSSHの設定をリロードします。
(6) ファイアーウォールの設定を確認しておく
ファイアーウォールのせいでリモート接続できない場合があります。下記のコマンドを実行すると、ポートの情報が出力されます。
22がALLOW Anywhereになっていなければ、下記のコマンドを実行してポートを開放する必要があります。
これでBioLinux8側の設定は終了です。
Windows10側の設定
(1) TeraTermを利用した接続
TeraTermを起動したら、ホストにBioLinux8のIPアドレスを指定して、OKをクリックします。
BioLinux8のIPアドレスが分からない場合は、BioLinux8側で下記のコマンドを実行すると、インターネット接続に関する情報が出てきます。
「inet addr」という項目があるので、それがパソコンのIPアドレスになります。
すると、SSH認証のウインドウに移行するので、BioLinux8にログインする際のユーザ名とパスフレーズ(パスワード)を入力します。さらに、「RSA/DSA/ECDSA/ED25519鍵を使う」にチェックを付けて、先ほどWindows10上で作成した秘密鍵を指定してOKをクリックします。
すると、BioLinux8の入ったパソコンにリモート接続できているはずです。
BioLinux/Ubuntu(3) CPU・メモリの負荷状態を視覚化するツール - sysmonitor
CentOSを利用していたとき、CPU・メモリの負荷状態を視覚化するツールが常駐しており、今どれくらいの負荷状態にあるか確認できて便利だなぁと感じていました。
UbuntuやBioLinuxではそのような可視化ツールは入っていないので、適当なツールを導入したいと思っていました。
そこで今回は、こちらのサイトで紹介されていた「sysmonitor」と呼ばれるCPU・メモリの負荷状態を可視化するツールを採用してみたいと思います。
(1) sysmonitorのインストール
下記の通りにコマンドを打ち込み、sysmonitorをインストール
(2) sysmonitorの設定
まず、sysmonitorを検索して起動させます。
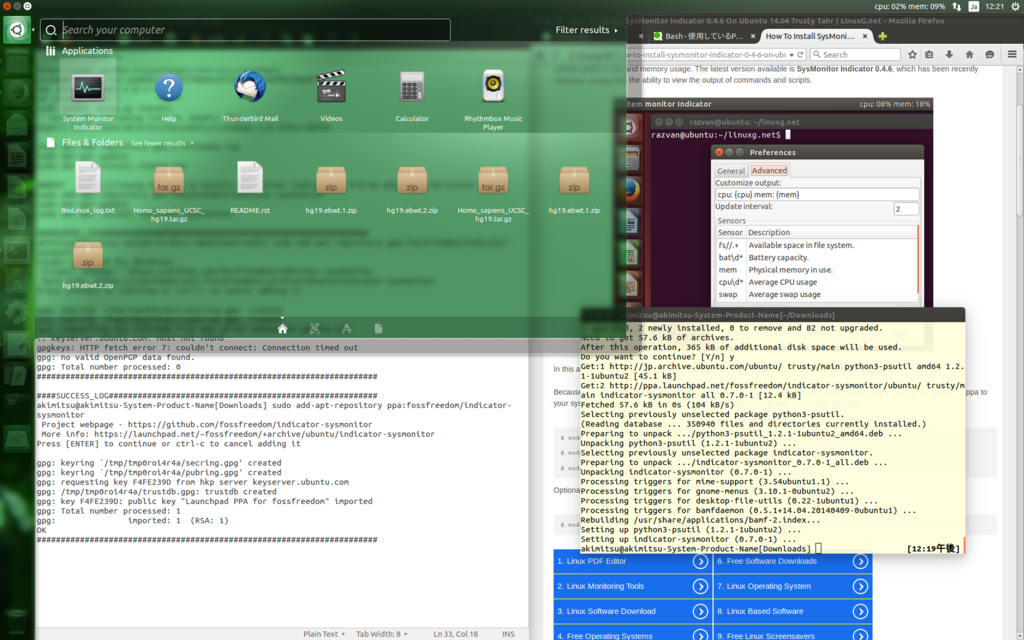
すると、右上のバーにCPUとメモリの使用状況がパーセンテージ(%)で表示されます。
普段、いちいちSystem Monitor indicatorを手動で起動させるのはだるいので、「cpu: XX% mem: YY%」と表示されているところを右クリックし、続いて「Preferences」をクリックします。すると、図のように設定画面がでてくるので、「Run on startup」にチェックをつけます。
こうすることで、起動時にSystem Monitor indicatorが自動的に立ち上がるようになります。
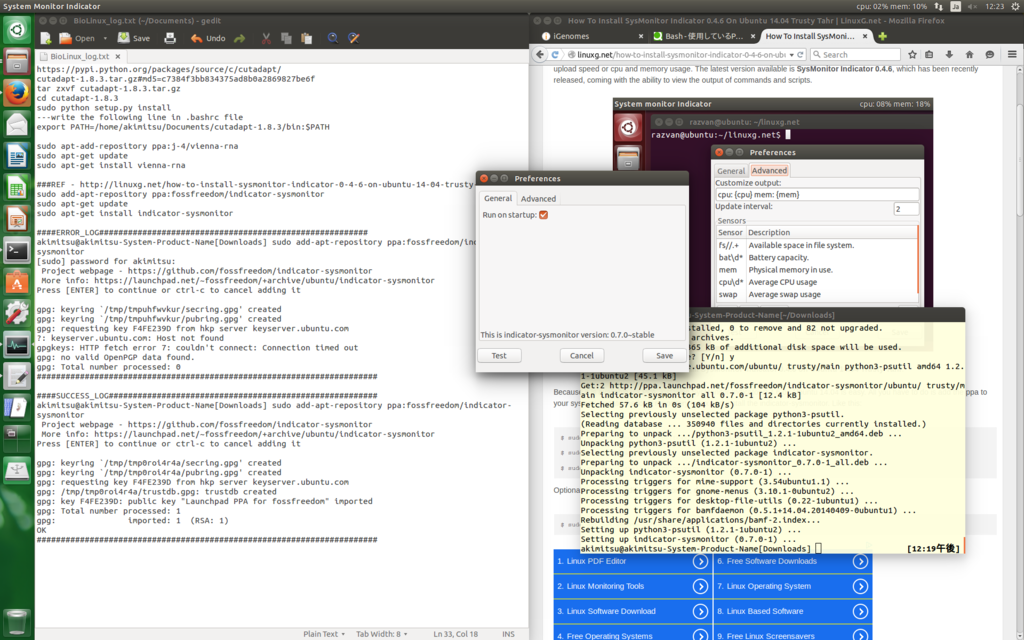
正直な話、System Monitor indicatorだと数値しか出ないので、継時的なCPU・メモリの使用状況をグラフとして表示させるGUIがほしいところです…。
BioLinux(2) BioLinux8の思わぬ落とし穴 - ログイン時のシェルは"bash"ではなく"zsh"
BioLinux8はLinuxディストリビューションであるUbuntu14.04をベースにつくられており、あらかじめバイオインフォマティクス解析用のソフトウェアがインストールされているから初心者向きだと聞きますが、思わぬ落とし穴があったので紹介します。
Ubuntu14.04では、ログインシェルはBash(/bin/bash)になっているのですが、BioLinux8では、zsh(/bin/zsh)がログインシェルに指定されています。
これが原因で、マニュアルでパスを通す際に問題に直面しました(PATHの設定については以下を参照)。
Ubuntu14.04では、ログイン時に~/.profileがまず読み込まれ、~/.profileから~/.bashrcが呼び出される仕組みになっていますが、BioLinux8ではシェルがデフォルトでzshになっているため、~/.bashrcが読み込まれません。
そのため、Ubuntu14.04のときのように、~/.bashrcファイルの末尾に以下のようなPATHを追加してもパスが通りません。
なので、ログイン時のシェルをzshではなく、bashに変更することにしました。下記のようにchshコマンドを使うことでシェルを変更することができます。
これでUbuntuと同様にパスの設定ができるようになりました。
補足
(1) 現在のシェルの確認
(2) BioLinux8に入っているシェルの種類を調べる
BioLinux(1) BioLinux8をデスクトップパソコンにインストールしてみた
BioLinux8は、Linuxのディストリビューションの1つであるUbuntu14.04をベースに作られたバイオインフォマティクス解析に特化したLinux OSです。
ぶっちゃけ、中身はUbuntuなんですが、NGS解析をはじめとする生物系のデータ解析に必要なソフトウェアがあらかじめインストールされています。environmentalomics.org
生物系のソフトウェアの中には、Perlなどの拡張モジュールやその他のソフトウェアに依存して動くものがあり、場合によってはそれらを自分でインストールしなければなりません。そのため、初心者にとってはソフトウェア1つをインストールするのも一苦労なんてこともあります(例えば、MEMEとか)。
そのため、あらかじめ必要なソフトウェアがインストールされており、初めから解析ができる環境を提供してくれるBioLinuxは「Linuxは初めて(>_<)」という初心者の方(自分を含め)におすすめなLinux OSだといえるでしょう。
ここでは、先週CentOSが入ったデスクトップパソコンからCentOSを消してBioLinux8を入れたときの備忘録を書きたいと思います。
1. BioLinux8インストール用のUSBメモリを準備する
OSを自分のパソコンにインストールしたことがある人ならわかると思いますが、どうやってパソコンにOSをインストールするかというと、CD/DVDやUSBメモリに入ったOSのデータをパソコンを起動させたときに呼び出すことでOSを自身のパソコンにインストールします。
そのために必要になるBioLinux8の入ったOSインストール用のUSBメモリをまず作ります。
必要なもの
- USBメモリ(4GB以上)
- インターネットに接続されたパソコン
(1) BioLinux8のダウンロード
まず、下記のURLからBioLinux8のisoファイルをダウンロードします。
Environmental Omics » Bio-Linux Installation
isoファイルへの直リンク
http://nebc.nerc.ac.uk/downloads/bio-linux-8-latest.iso
(2) UNetbootinのダウンロード・インストール
実は、ダウンロードしたBioLinux8のisoファイルをそのままUSBメモリにコピーしても、OSインストール用のUSBメモリとして機能しません(パソコン起動時にOSのインストールが開始されません…)。
ブートできるUSBメモリを作成するために、UNetbootinと呼ばれる専用のソフトを使います。下記のサイトからソフトをダウンロードして、自分のパソコンにインストールしましょう。
UNetbootin - Homepage and Downloads
(3) isoファイルをUSBファイルに書き込む
買ってきたUSBメモリをパソコンに指し、UNetbootinを起動します。UNetbootinのサイトに書いてあるとおり、「Diskimage」にチェックを入れ、先ほどダウンロードしたBioLinux8のisoファイルを指定してください。そうしたら、「OK」ボタンをクリックしisoファイルをUSBメモリに書き込みましょう。
ここまで出来たら、BioLinux8をインストールするためのUSBメモリの完成です。
2. BIOSの設定(USBメモリが一番先に読み込まれるようにする)
用意したデスクトップパソコンにBioLinux8をインストールする前にパソコン自体の設定をしなければいけません。
パソコンが起動した時、ハードディスクやCD/DVD、USBがどの順番で読み込まれるか順番があらかじめ決められています(下図では、CD/DVD→ハードディスク→USBの順)。
今回はUSBメモリ経由でOSをインストールするので、パソコン側がUSBを一番最初に読み込むように設定する必要があります(BIOSの設定)。
BIOSの設定はマザーボードのメーカーによって異なるので、ここでは例としてASUSのBIOS設定について示したいと思います。
「Boot Priority」中で青くなっているのがUSBです。
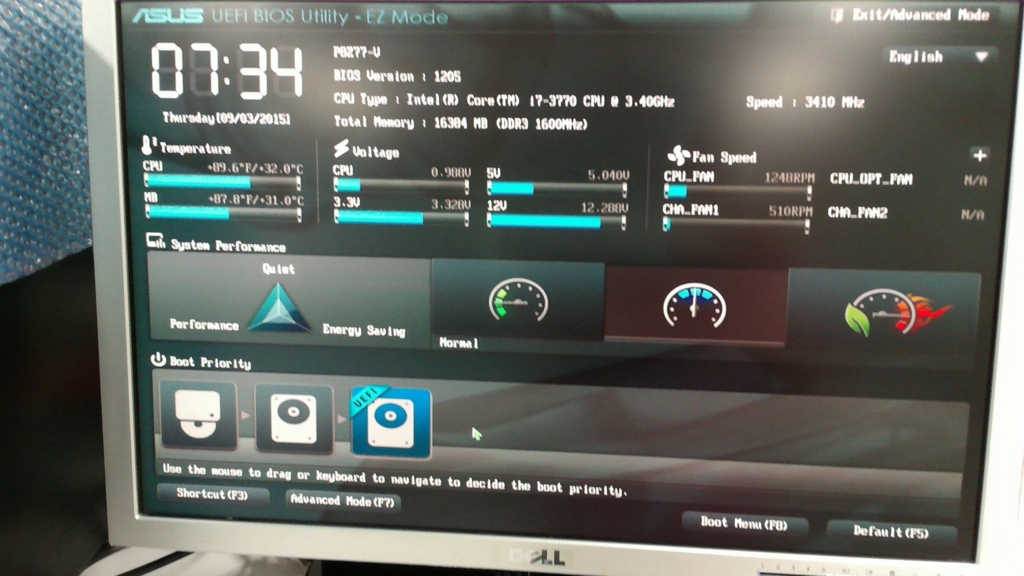
アイコンをドラック&ドロップで一番左に持ってきます。これでパソコン起動時にUSBが一番最初に読み込まれるように設定されました。
あとは、右上の「終了/アドバンスト」ボタンをクリックしていったんパソコンをシャットダウンします。

3. BioLinux8のインストール
1で作成したUSBメモリをパソコンに挿した後、パソコンを起動させます。
すると、以下のような画面になるので、「Install Bio-Linux 8」を選択して「Enter」を押します。
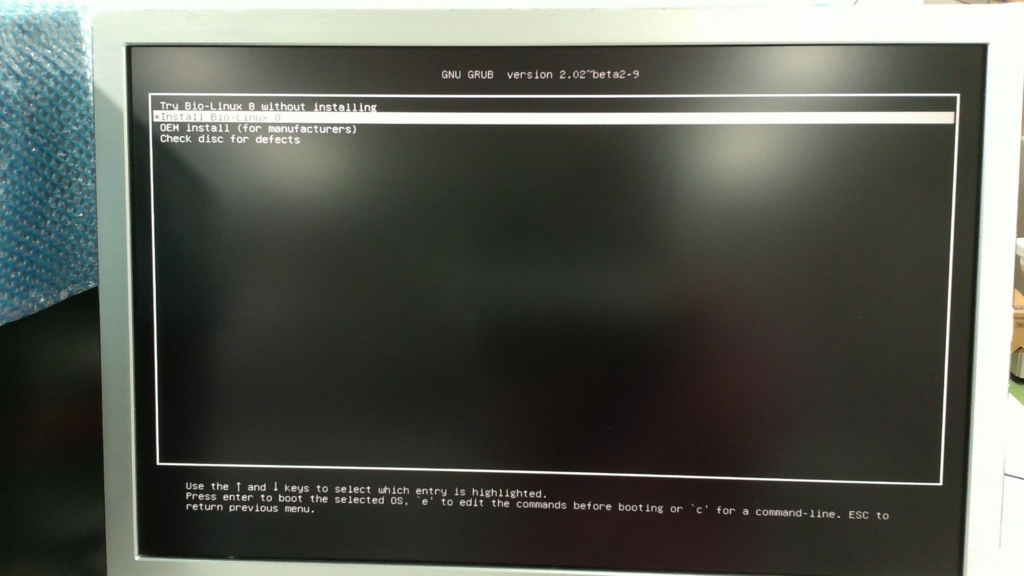
Bio-Linuxのロゴが出現します。

Welcomeの文字が出てきます。まずは言語の設定でデフォルト通りに「English」を選択し(日本語を選択するとうまくBioLinux8がインストールされない可能性があります)、「Continue」をクリックします。
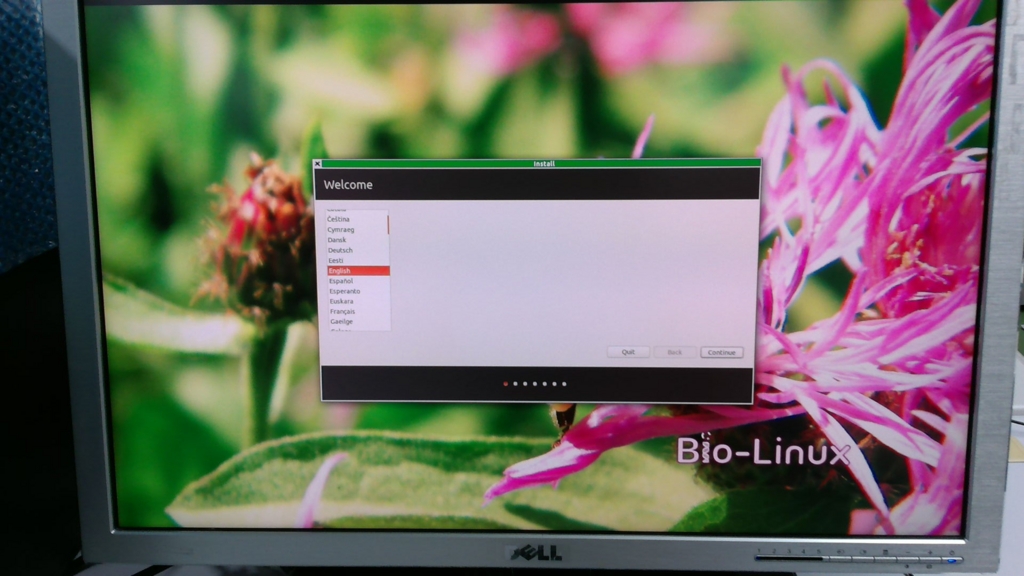
何も考えずに「Continue」をクリック。
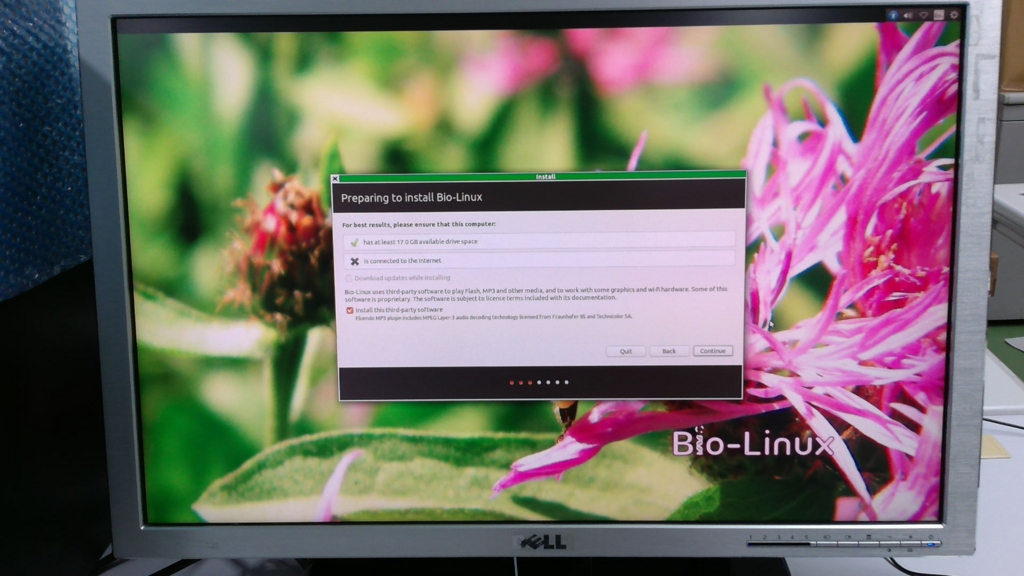
一番上にチェックが付いていることを確認して、「Continue」をクリック。
一番上を選択すると、ハードディスク内に書き込まれた情報をすべて消去し、BioLinux8がインストールされます。今回の場合、CentOSが入っていたのでこれを選択することによりCentOSが消えてBioLinux8が代わりにインストールされます。
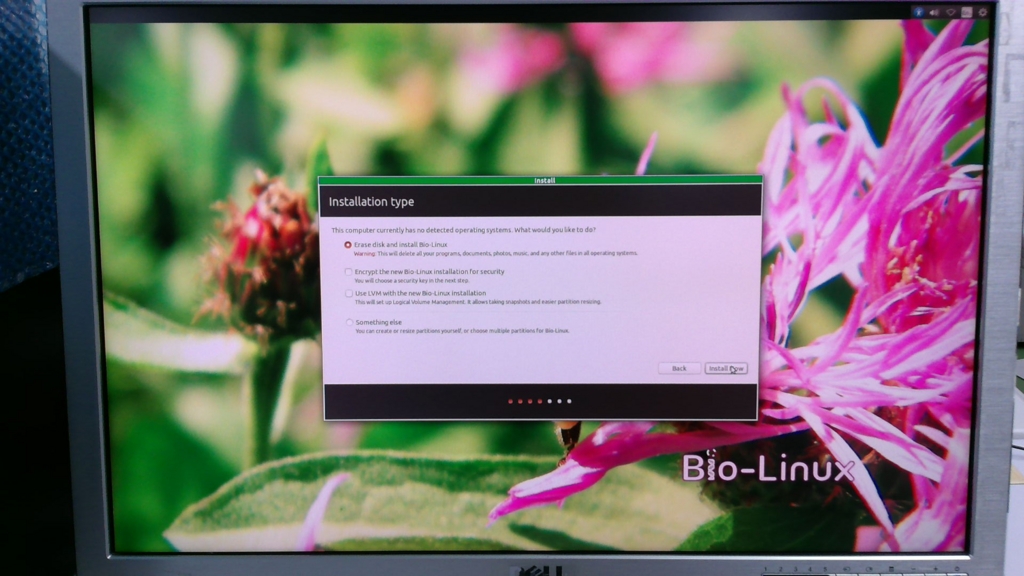
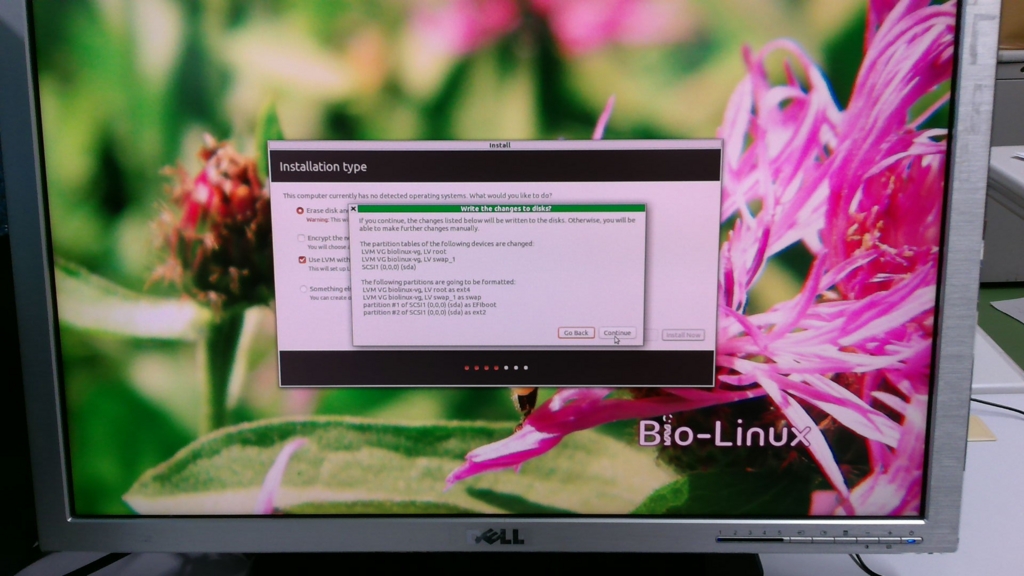
ハードディスクのパーティション分けに関して聞かれているよう。とりあえず、何も考えずに「Continue」をクリック。
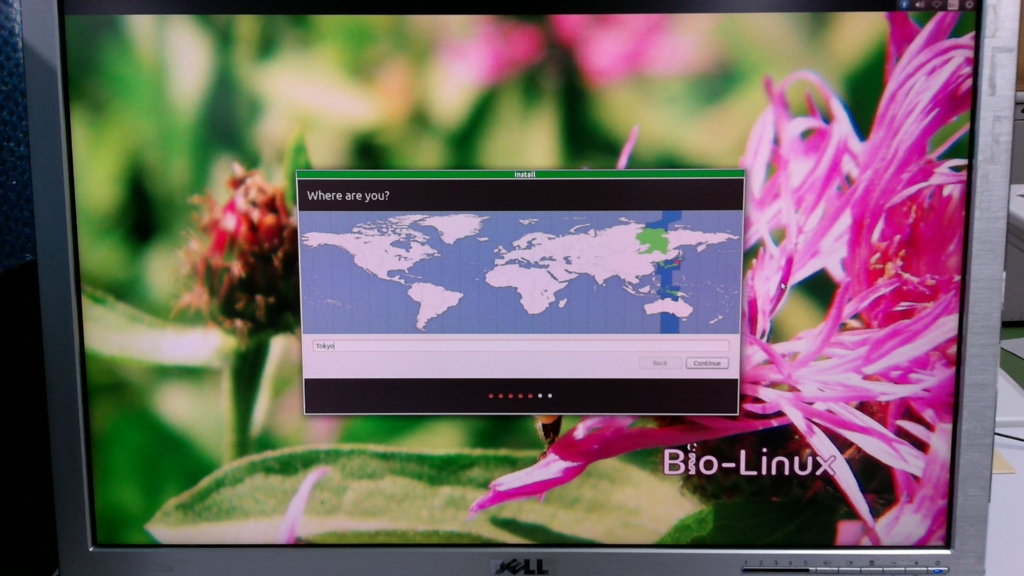
所在地はどこだと聞かれたので、地図上で「Tokyo」を選択して、「Continue」をクリック。
キーボードの設定を聞いてくるので、日本語のキーボードを選択(英語のキーボードを使っている人は英語のを選択)。

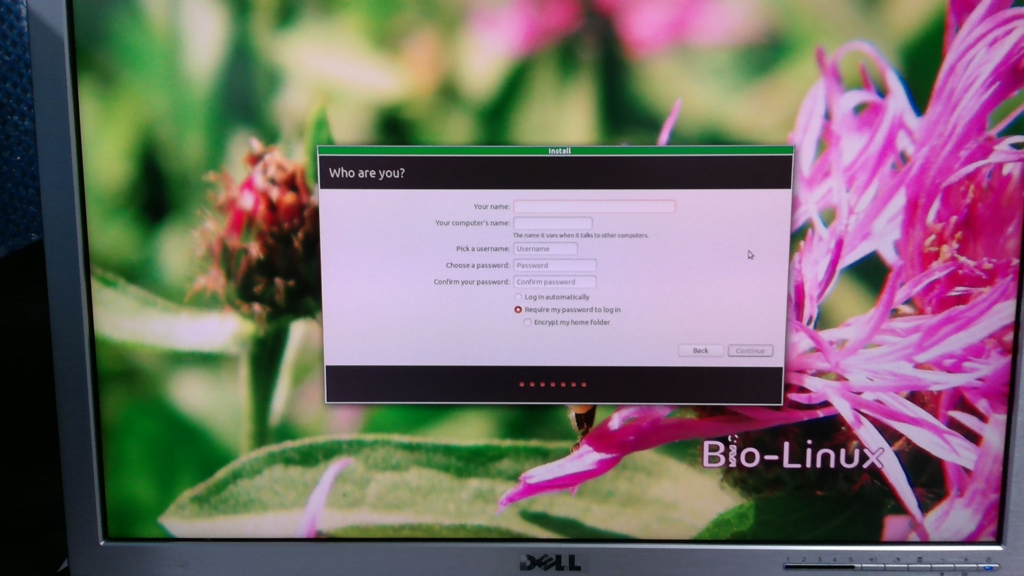
「username」と「passward」の設定。この2つはログイン時に必要になるので、忘れないようにしておきましょう。また、「Log in automatically」を選択しておくと、パソコン起動時に自動でログインするように設定できます。
インストールが始まりました。
(私がやったときは謎のエラーが起こりました。CentOSは消えてしまい、BioLinuxもインストールされず。ラボのパソコンが”ただのでかい箱”になってしまい、青ざめました…。その時は、ハードディスクが一番最初に読み込まれるようにBIOSの設定を行い、パソコンを再起動。さっきとは少し違う画面で、UNetbootinの設定画面が出てくるのですが、Install Bio-Linux8を選択しました。再びBioLinux8のインストールを行ったところ、無事にインストールされました。なんでエラーが出たのかはよくわかりません…。)
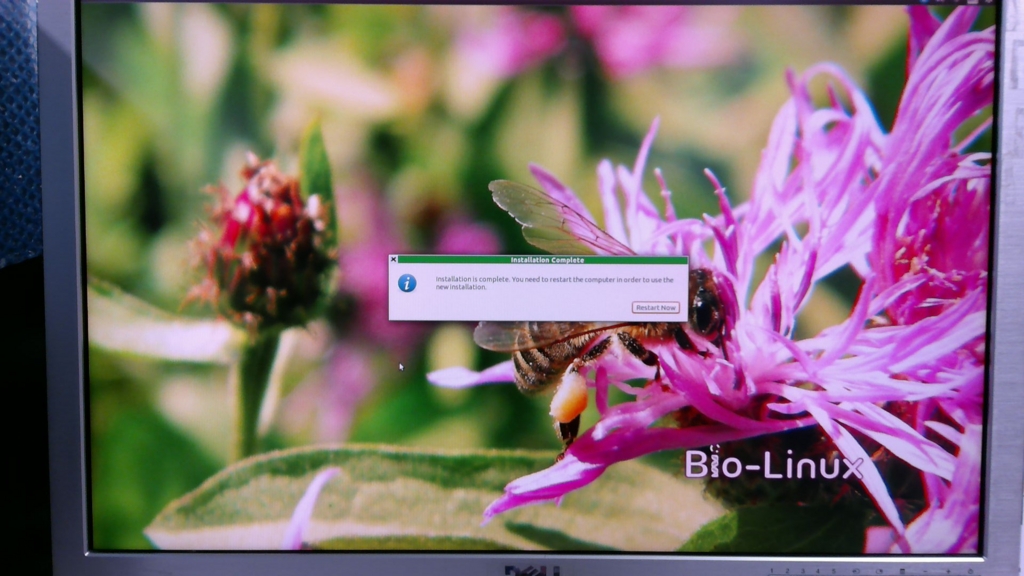
無事に、インストール完了!
以上でBioLinux8のインストールは完了です。思っていたより簡単な作業ですね(エラーさえでなければ…)。
Python(1) PyCallGraphを利用したコードプロファイリングの可視化
最近Pythonを使って、特定のmicroRNAが結合するSeed配列をmRNAの配列中から探索して、さらに種間保存性や配列の特徴を解析するスクリプトを思いつくままに書いたのですが、
実行してみて、そのスクリプトがやたら遅いことに気づきました(-_-;)
「1つのmiRNA vs すべてのmRNA」のペアで3時間以上かかる…。すべてのmiRNAとすべてのmRNAの組み合わせを実行したら、いったい計算に何ヶ月かかるんだろうってレベルです。
スクリプトはこちら。
そこで、スクリプトのどの部分が計算上ボトルネックになっているか調べてみることにしました。POSTDに掲載されていたPythonにおけるコードプロファイリングのお話があったので早速試してみました。
上記の記事でも説明されているように、 PythonにはcProfile (pythonのcProfileを使ってみる。 - malibu-bulldogの日記)と呼ばれるプロファイリングツールが用意されています。しかし、実行してみるとログがだぁーと出力されてくるだけで、何を言っているのか直感的に理解できません…。
そこで、PyCallGraphの出番です。PyCallGraphは上記のcProfileの出力情報を視覚化すてくれます。もちろん、ビルトインモジュールではないので、自身でインストールを行う必要があります。
(1) PyCallGraphのインストール
PythonのモジュールであるPyCallGraphとGraphvizをそれぞれインストールします(今回はPython3のモジュールとしてインストールしました)。
また、グラフを描写するために必要となるGraphviz自体がインストールされていない場合は、合わせてインストールする必要があります。当たり前ですが、GraphvizがないとPyCallGraphが使えません(実行するとエラーが返ってくる)。
(2) PyCallGraphの実行
下記の通り、pycallgraphからスクリプトを呼び出すだけでOKです。簡単ですね。
(3) 実行結果
結果としてpycallgraph.pngというファイルがカレントディレクトリに生成されます(この時、同じ名前のファイルが既に存在しているとエラーを起こす原因になるかもしれません)。 出力された画像ファイル(.png)の一部を切り取ってきたのが下図になります。
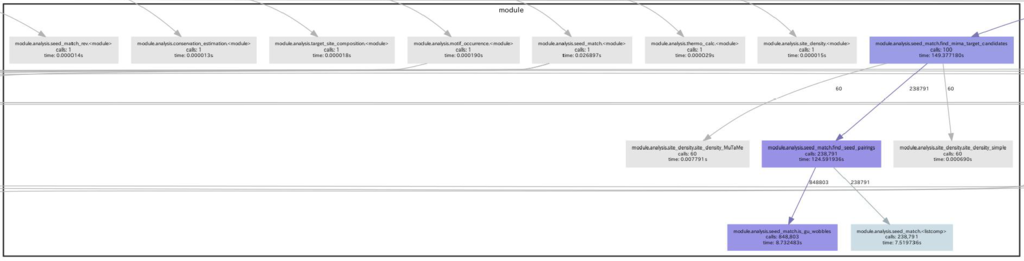
細かくて字がつぶれていますが、「module.analysis.seed_match.find_mirna_target_candidate」モジュールとそれに従属するモジュール群が青色になっています(それ以外は灰色)。
これは、これらのモジュール上で計算に時間がかかっていることを示しています(計算上のボトルネック)。以上の結果から、seed配列の探索に時間がかかっていることが判明しました(計算時間のほとんどがseed配列の探索に割かれている…)。
このようにPyCallGraphを利用したコードプロファイリングを行うことで、スクリプトのどの個所に計算上のボトルネックが存在しているのか視覚的に把握することができます。
NGS関係(2) Adapter sequence letter for illumina customers
smallRNA-seqやCLIP-seqなどシークエンスされたリードにアダプター配列が入っています。これは、smallRNAやRNA結合タンパク質に結合しているRNA配列の長さが短いため、アダプター配列まで読まれているためです。
そのため、例えばilluminaが出している「TruSeq Small RNA Sample Prep Kit」を使ってシークエンスしたデータに関しては、下記のURLから、illuminaが提供しているアダプター配列に関する情報を入手してアダプター配列をトリミングする必要があります。
また、CLIP-seqに関しては、PAR-CLIPであれば基本的に下記の論文に記載されているアダプター配列を使用しているようです。
データベース関係(1) miRBase Tracker - 地味に便利なmiRBaseのバージョン比較データベース
miRBaseは言わずと知れたmiRNAのデータベースですが更新のペースが早く(近年では年一回ペースと落ち着いている)、登録されているmiRNAのレコードが変更されたり、削除されることもしばしばです。
アクセッション番号はもちろん変更されることなく普遍的な番号になっているのですが、miRNAの名称自体はころころ変更されています。
これによってどのような問題が生じるかというと、例えば、ある論文ではmiRBase v16時点でのmiRNA名称を使用していて、miRBase v20での正式名称はどうなっているを簡単に参照することができないとか…。
それ以外にも、次世代シーケンサーのデータでmiRBase v16で集計されたものと、miRBase v19で集計されたものを比較したい時に、どのmiRNAのレコードが削除されていたり変更されているか確認しておきたいなんてことはありませんか(そのくらい自分で比較して調べろって話かな)。
そんなちょっとした問題を解決してくれるのが、
というデータベースです。
論文はこちら。
以下は、データベースの詳細になります。

(1)特定のmiRNAに関して、どのバージョンで名称などの変更がなされたか視覚的に確認することができます。
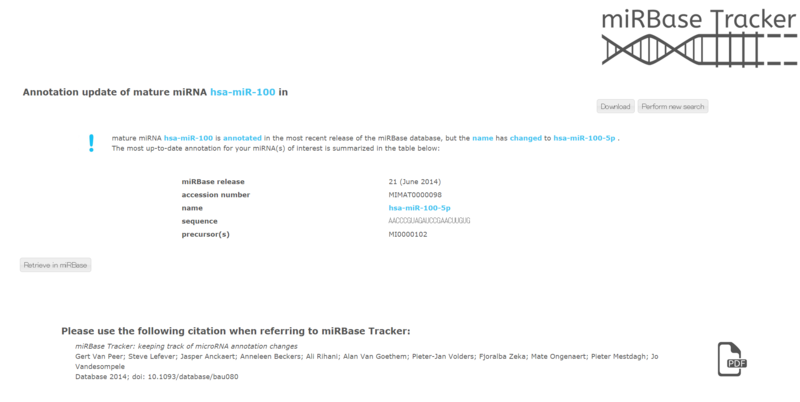
(2)最新の情報を確認することができます。旧バージョンから変更されている点が一目瞭然。
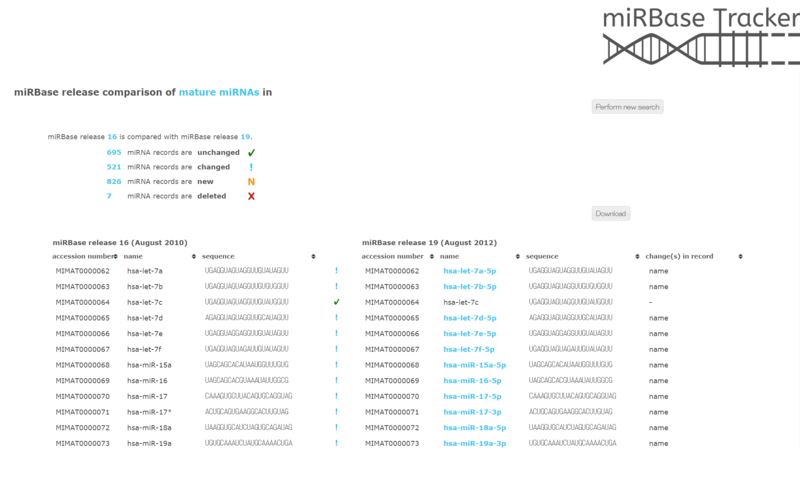
(3)miRBaseに登録されているmiRNAのデータをすべて比較した対応表。2つの異なるバージョン間の比較ができます。また、その情報をダウンロードすることも可能です。おそらく、一番使える情報なのでは。
こんなデータベースもあるんですね。GencodeやRefSeqなどのアノテーション情報もこのように見やすい形でバージョン間の比較をしてもらえると嬉しいなぁ。